Ablesen der DNA: Wie Transkription das Genom formt
18.02.2022
LMU-Wissenschaftler zeigen, dass ein grundlegender Prozess des Lebens anders abläuft, als bisher weithin angenommen wurde.
18.02.2022
LMU-Wissenschaftler zeigen, dass ein grundlegender Prozess des Lebens anders abläuft, als bisher weithin angenommen wurde.
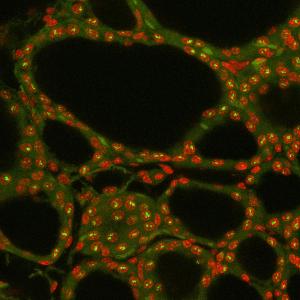
(Zellkerne rot, RNA-Signal grün). Bild: I. Solovei
Damit die in der DNA gespeicherten genetischen Informationen von der Zelle umgesetzt werden können, müssen sie von bestimmten Enzymen – sogenannten RNA-Polymerasen – abgelesen und in RNA-Moleküle übersetzt werden. Dieser grundlegende Vorgang des Lebens wird als Transkription bezeichnet. Sie findet im Inneren des Zellkerns statt, wo sich die aktivsten Gene befinden. Ein Team um Dr. Irina Solovei vom Biozentrum der LMU hat nun in Kooperation mit Prof. Leonid Mirny (MIT, Cambridge, USA) die räumliche Organisation der Transkription und das Verhalten der abgelesenen Gene untersucht und einen Mechanismus aufgedeckt, der einem weit verbreiteten Konzept widerspricht.
Trotz großer Fortschritte beim Verständnis der molekularen Mechanismen der Transkription ist bisher nur wenig über die räumliche Struktur einzelner Gene beim Ablesevorgang bekannt. Nach der gängigsten Hypothese sind die RNA-Polymerasen in einer Art stationärer Transkriptionsfabrik zusammengeschlossen, der sich aktivierte Gene nähern und durch sie hindurchbewegen. Allerdings ist bekannt, dass in speziellen Riesenchromosomen die Start- und Endstellen der Transkription im Raum fixiert sind und die exprimierten Sequenzen in einer Schleife verlaufen – hier bewegen sich die RNA-Polymerasen also entlang der Gene.
„Ich habe immer vermutet, dass es bei Eukaryoten nicht zwei grundsätzlich verschiedene Mechanismen der Transkription geben kann. Damit stellt sich die Frage: Was bewegt sich und was bleibt stationär?“, sagt Solovei. Die Biologin geht davon aus, dass die Auflösung moderner Lichtmikroskope in den meisten Fällen nicht gut genug ist, um die in den Riesenchromosomen beobachteten Transkriptionsschleifen sichtbar zu machen. Gerade häufig exprimierte Gene sind bei Eukaryoten oft sehr kurz und unterschreiten die mögliche Auflösung – und große Gene werden in der Regel nicht stark exprimiert und sind daher für Transkriptionsstudien ungeeignet.
Soloveis Team identifizierte daher in Mäusezellen mehrere lange und stark exprimierte Gene, die unter dem Lichtmikroskop dargestellt werden können, und untersuchte deren Struktur. Die Wissenschaftlerinnen und Wissenschaftler fanden, dass sich diese Gene während der Transkription auffalten, wobei sich ihre Enden voneinander entfernen und stark in den Kernraum ausdehnen. Dabei bilden sie Transkriptionsschleifen, die denjenigen der Riesenchromosomen ähneln.
Wie die Forscher nachwiesen, bewegen sich die RNA-Polymerasen entlang dieser Schleifen und transportieren entstehende RNA-Moleküle, die dabei einen für Eukaryoten charakteristischen Prozess der RNA-Reifung durchlaufen. „Die Form und die Ausdehnung der Transkriptionsschleifen von Trägerchromosomen deuten darauf hin, dass es sich bei den Transkriptionsschleifen um starre Strukturen handelt“, sagt Solovei. Die Autoren vermuten, dass hochexprimierte Gene steif werden, weil sie dicht mit mehreren entstehenden RNAs besetzt sind, die in Kombination mit RNA-bindenden Proteinen voluminöse Partikel bilden.
Unterstützt wird diese Hypothese auch durch eine Polymermodellierung exprimierter Gene, die von Mirnys Gruppe durchgeführt wurde. Durch die Simulation des Verhaltens von transkribierten Genen konnten die Forscher zeigen, dass die modellierten Gene erst ab einer bestimmten Steifigkeit biologische Beobachtungen widerspiegeln. „Das Auffälligste für mich ist die schiere Größe der Transkriptionsschleifen – bis zu 10 Mikrometer", sagt Mirny. „Es scheint, dass ein einziges langes Gen sich entfalten und den größten Teil des Zellkerns durchqueren kann."
„Die Ergebnisse der Studie belegen“, so Prof. Heinrich Leonhardt, Inhaber des Lehrstuhls für Humanbiologie und BioImaging, „dass die Mikroskopie auch in unserer von der Molekularbiologie dominierten Zeit ein wichtiges und leistungsfähiges Instrument zur Untersuchung grundlegender biologischer Fragen bleibt.“
Susanne Leidescher, Johannes Ribisel, Simon Ullrich, Yana Feodorova, Erica Hildebrand, Alexandra Galitsyna, Sebastian Bultmann, Stephanie Link, Katharina Thanisch, Christopher Mulholland, Job Dekker, Heinrich Leonhardt, Leonid Mirny, Irina Solovei: Spatial organization of transcribed eukaryotic genes. Nature Cell Biology 2022