Biomoleküle: Nanometermaß auf dem Prüfstand
27.03.2023
Forscher haben im Zuge einer internationalen Vergleichsstudie ein Verfahren zur Untersuchung dynamischer Proteinstrukturen erfolgreich getestet und validiert.
27.03.2023
Forscher haben im Zuge einer internationalen Vergleichsstudie ein Verfahren zur Untersuchung dynamischer Proteinstrukturen erfolgreich getestet und validiert.

Laser im Labor von Thorben Cordes | © Cordes / LMU
Die exakte Vermessung von Biomolekülen kann entscheidend dazu beitragen, grundlegende Prozesse des Lebens besser zu verstehen. Ein Team um die LMU-Wissenschaftler Prof. Thorben Cordes und Prof. Don C. Lamb sowie Prof. Claus Seidel von der HHU Düsseldorf und Dr. Anders Barth an der TU Delft hat nun in einer groß angelegten Vergleichsstudie, an der 19 Labore aus aller Welt beteiligt waren, eine Methode zur Vermessung von Biomolekülen auf ihre Genauigkeit und Vergleichbarkeit geprüft. Die Ergebnisse wurden nun im Fachmagazin Nature Methods veröffentlicht.
Proteine sind die Grundbausteine des Lebens. Jedes Tier, jede Pflanze und jeder Mikroorganismus ist daraus zusammengesetzt und funktioniert nur aufgrund zahlloser komplexer Prozesse, die durch das Zusammenspiel verschiedener Proteine gesteuert werden. Kein Wunder also, dass die Wissenschaft ein großes Interesse daran hat, diese Multitalente der Biochemie besser zu verstehen.
Das Problem dabei ist allerdings, dass man sie natürlich nicht einfach mit einem Zollstock vermessen kann. Forschende bedienen sich deshalb eines ganzen Werkzeugkoffers verschiedener Untersuchungsmethoden, um ein genaueres Bild davon zu erhalten, wie Proteine aussehen, sich verhalten und wie sie funktionieren.
Die Einzelmolekül-FRET-Analyse ist besonders dafür geeignet. Sie nutzt den sogenannten Förster-Resonanzenergietransfer (FRET), bei dem Energie eines angeregten Farbstoffs strahlungsfrei auf einen zweiten übertragen wird. Indem künstlich Farbmoleküle in die untersuchten Biomoleküle eingebracht werden, kann man so sehr kleine Abstände im Sub-Nanometerbereich messen.
Für die Messung von Abständen zwischen verschiedenen Molekülen funktioniert das bereits recht gut. Auch die Struktur von DNA-Strängen lässt sich schon ziemlich verlässlich untersuchen. Im Vergleich zu DNA ist die Sache bei Proteinen jedoch um einiges kniffliger: Sie sind vielfältiger und vor allem beweglicher und daher viel schwerer zu untersuchen.
Trotzdem gelang es den Forschern in der Studie, das Verfahren nun auch für bewegliche Proteine erfolgreich so zu etablieren, dass sich mit der Methode reproduzierbare und genaue Ergebnisse erzielen lassen. So konnten sie damit nicht nur winzige Abstände innerhalb der Eiweißkomplexe feststellen, sondern auch strukturelle Unterschiede beobachten, wenn die Proteine ihre Form veränderten.
Bisher waren viele Kollegen und Kolleginnen in der Strukturbiologie skeptisch, ob die Analyse von Proteinen mittels FRET überhaupt zu reproduzierbaren Ergebnissen führen kann und auch wie die Ergebnisse zu interpretieren sind, wenn sich die Proteine bewegen. Diese Zweifel konnten wir nun ausräumen.Thorben Cordes
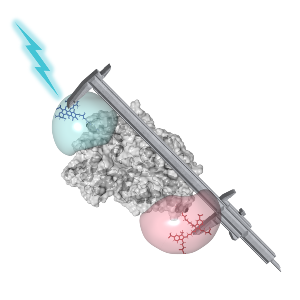
Visualisierung der Vermessung von Proteinen mit der FRET-Methode | © Simon Wanninger (AK Lamb, LMU) & Christian Hanke (HHU)
Solche Strukturveränderungen konnten die beteiligten Labore auf weniger als einen Nanometer genau und zwar auf Zeitskalen von weniger als einer Millisekunde bestimmen. Diese erstaunliche Präzision zeigt, dass auch dynamische Proteinsysteme mittels FRET reproduzierbar erfasst werden können.
„Bisher waren viele Kollegen und Kolleginnen in der Strukturbiologie skeptisch, ob die Analyse von Proteinen mittels FRET überhaupt zu reproduzierbaren Ergebnissen führen kann und auch wie die Ergebnisse zu interpretieren sind, wenn sich die Proteine bewegen“, sagt Thorben Cordes. „Diese Zweifel konnten wir nun ausräumen. Damit zeigen wir aber auch, wie winzig und schnell Proteinbewegungen sein können, sodass wir sie mit FRET noch beobachten und quantifizieren können.“
Damit ist der Werkzeugkasten der Strukturbiologie um ein weiteres vielseitiges und zuverlässiges Instrument reicher geworden, meinen die Forscher. Sie hoffen, dass die so generierten Daten auch die Aussagekraft KI-basierter Vorhersagen verbessern und somit das Verständnis dynamischer Prozesse bei Proteinen weiter vorantreiben.
Ganesh Agam, Christian Gebhardt, Milana Popara et al. Reliability and accuracy of single-molecule FRET studies for characterization of structural dynamics and distances in proteins. Nature Methods (2023).